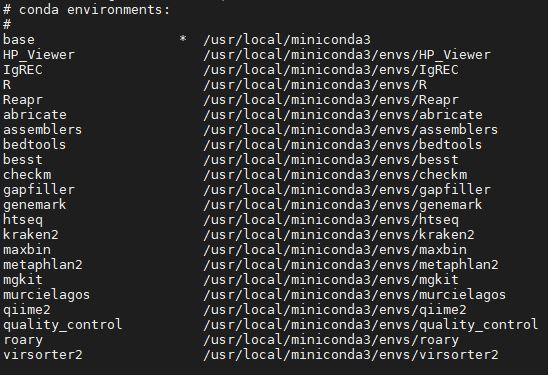
Ambientes miniconda
Aquí encontrarás información de los programas y sus versiones que se encuentra alojados en el servidor Biocluster2, y que son gestionados a través del gestor de paquetes miniconda3.
En la siguiente imagen se visualiza un listado de los ambientes existentes hasta el día de hoy. Además, se proporciona una pequeña descripción de los programas alojados en cada ambiente.
Ambientes
Base (Versión)
bamtools = (2.5.1) |
blast = (2.5.0) |
bowtie2 = (2.2.5) |
bwa = S(0.7.17) |
fastqc = (0.11.9) |
hmmer = (3.3.2) |
perl = (5.32.1) |
python = (3.9.7) |
r-base = (3.6.1) |
samtools = (1.6)
HP_Viewer
IgREC
biopython = (1.76) |
numpy = (1.12.1) |
python = (2.7.12) |
scipy = (0.18.1)
R
Abricate
blast = (2.12.0) |
bowtie2 = (2.2.5) |
bwa = S(0.7.17) |
fastqc = (0.11.9) |
hmmer = (3.3.2) |
perl = (5.32.1) |
python = (3.9.7) |
r-base = (3.6.1) |
samtools = (1.6)
qiime2
assemblers
bowtie = (1.3.1) |
bowtie2 = (2.4.5) |
perl = (5.32.1) |
python = (3.9.7) |
samtools = (1.15.1) |
spades = (3.14.1) |
trimmomatic = (0.39) |
trinity = (2.8.5) |
velvet (2.8.5)